PDB ファイルの前処理
データバンクから入手した PDB ファイルや PDBx/mmcif ファイルファイルは実験結果を表現したものです.
原子の重複や欠落が見られたり,目的とするモデリングに不要な原子が含まれていたりします.
目的に応じた形に整形するための一助として,クリーンアップ(水分子,重複原子の除去),ペプチドに水素付加,ペプチドから水素除去の機能を作成しました.
目次(ページ内リンク)
クリーンアップ:水分子(遊離の酸素原子)および重複している原子を削除します
水素付加/水素除去:ペプチドに水素原子を付加/ペプチドから水素原子を除去します
クリーンアップ
PDB ファイルには,異常に接近した同一の元素の原子が記述されている場合があります.
原子間距離が共有結合半径の 1/4 未満であれば,実験の都合で重複した原子だと推定し,1 個の原子を残して他は削除します.
また,PDB ファイルには水分子(遊離の酸素原子)が含まれている場合があります.これも除去します.
クリーンアップのモデル
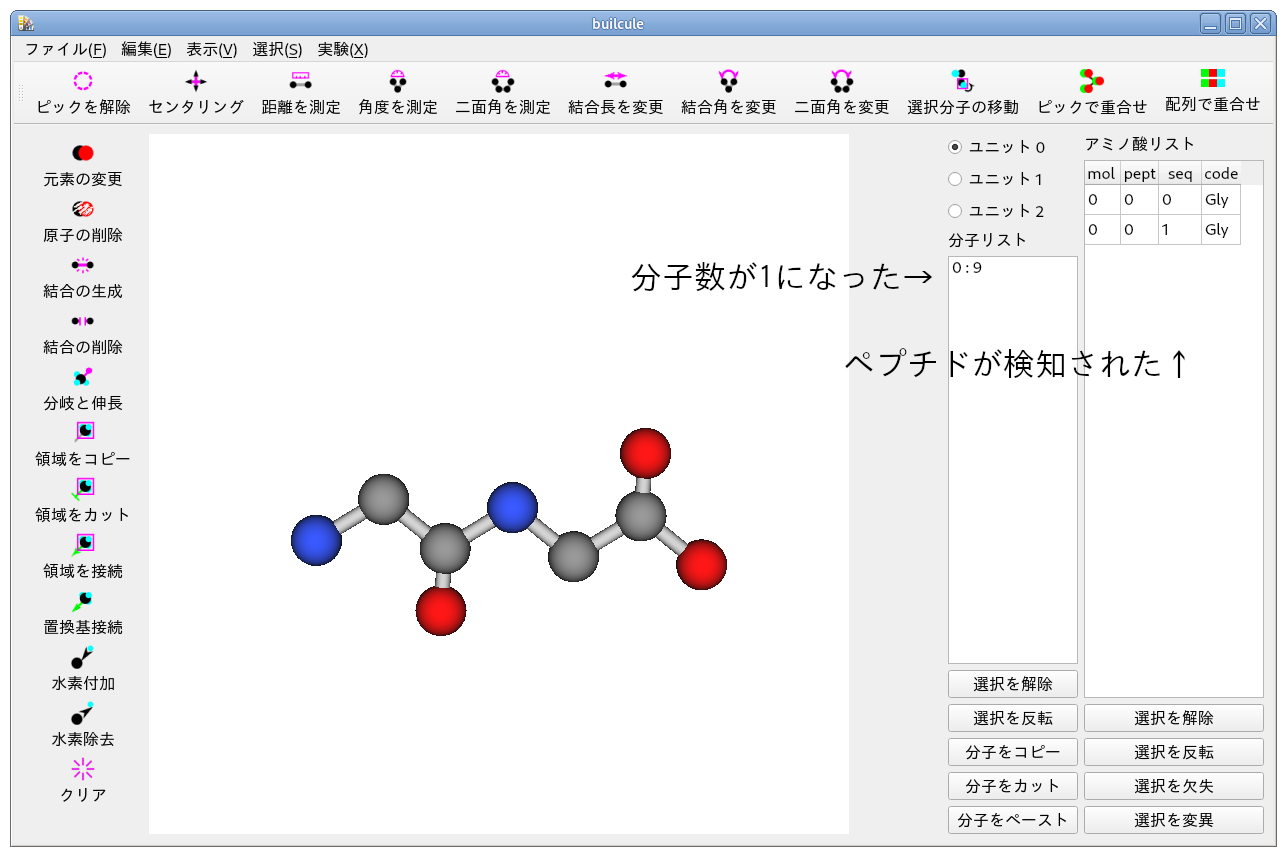
画像は,クリーンアップ機能を紹介するために作成したモデル系です.
ジグリシンの α-炭素原子の一つを重複させ,遊離の酸素原子を 4 個加えたものです.
炭素原子が重複しているため,ペプチドが検知されていません.
クリーンアップ後

画像は,メニューバーで [編集(_E)]-[クリーンアップ(_A)] としたところです.
分子数が 1 になり,ペプチドが検知されました.
水素付加/水素除去
ペプチド(タンパク質)に水素付加したり,ペプチドから水素を除去したりする方法を紹介しています.
X 線回折法で得られる構造には,水素が含まれないので,それを補填するための機能です.
オプション
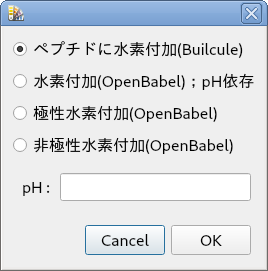
垂直ツールバーの![]() 水素付加ボタンをクリックすると画像に示すオプション選択ウィンドウが現れます.
水素付加ボタンをクリックすると画像に示すオプション選択ウィンドウが現れます.
![]() 水素除去ボタンでも同様です.
水素除去ボタンでも同様です.
ここでは,[ペプチドに水素付加(Builcule)] オプションを紹介します.
他は OpenBabel の機能です.
水素付加は,電荷した型を基本とします.
例えば,アミノ基は -NH3,カルボキシル基は-COO とします.
Arg 側鎖のグアニジウム基も正電荷を 1 個持つように付加します(水素はすべて sp3 型としています).
His 側鎖のイミダゾール基(pKa 6.0)も電荷型として処理します.すなわち,イミダゾール環の 2 個の窒素にはともに水素を付加します.
ヒドロキシプロリン以外の異常アミノ酸の側鎖や遊離アミノ酸には何もしません.
水素除去されたペプチド
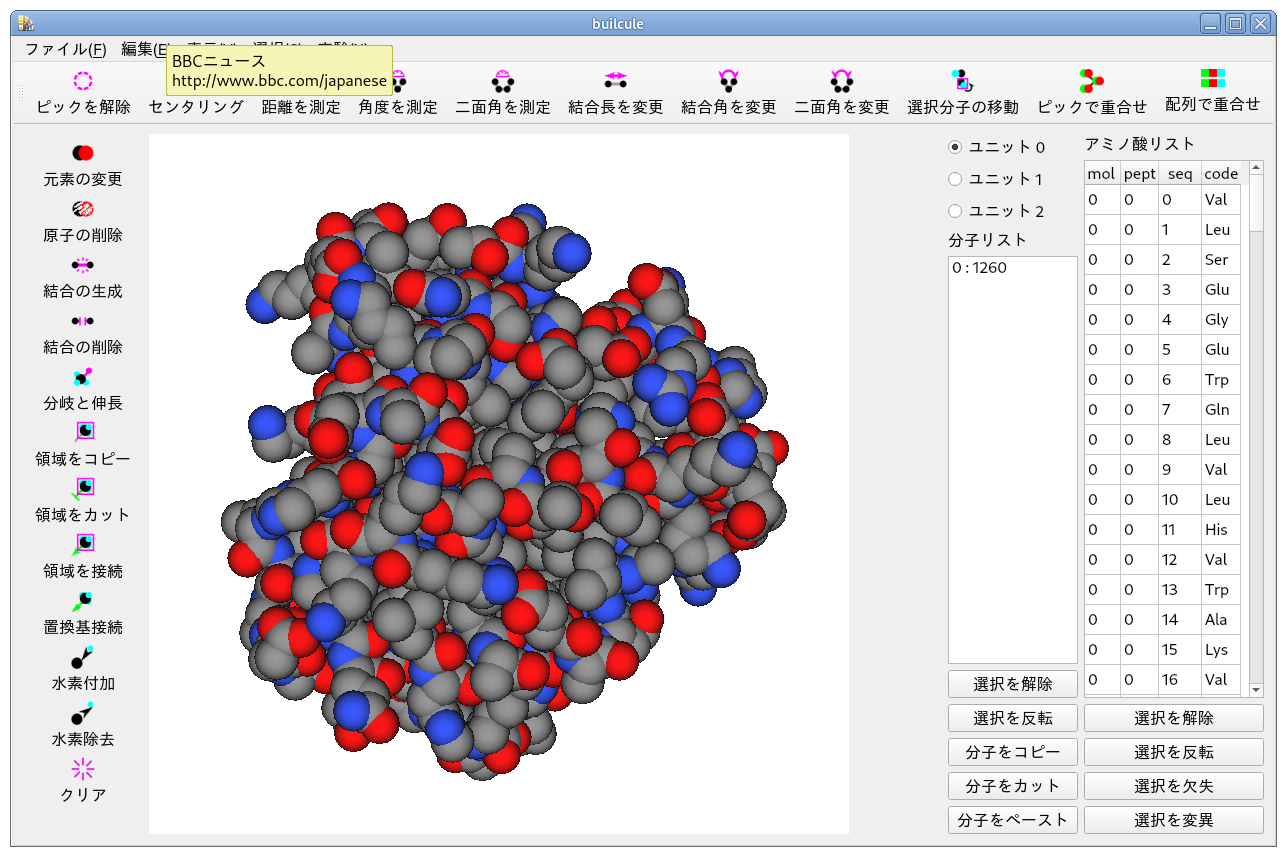
画像は,ある PDB ファイルを開いたところです.
[表示(_V)]-[空間充填(_S)]で表示しています.
このタンパク質は X-線結晶回折法で決定された構造なので,水素が含まれていません.
垂直ツールバーの![]() 水素付加ボタンをクリックすると下の状態になります.
水素付加ボタンをクリックすると下の状態になります.
水素付加されたペプチド
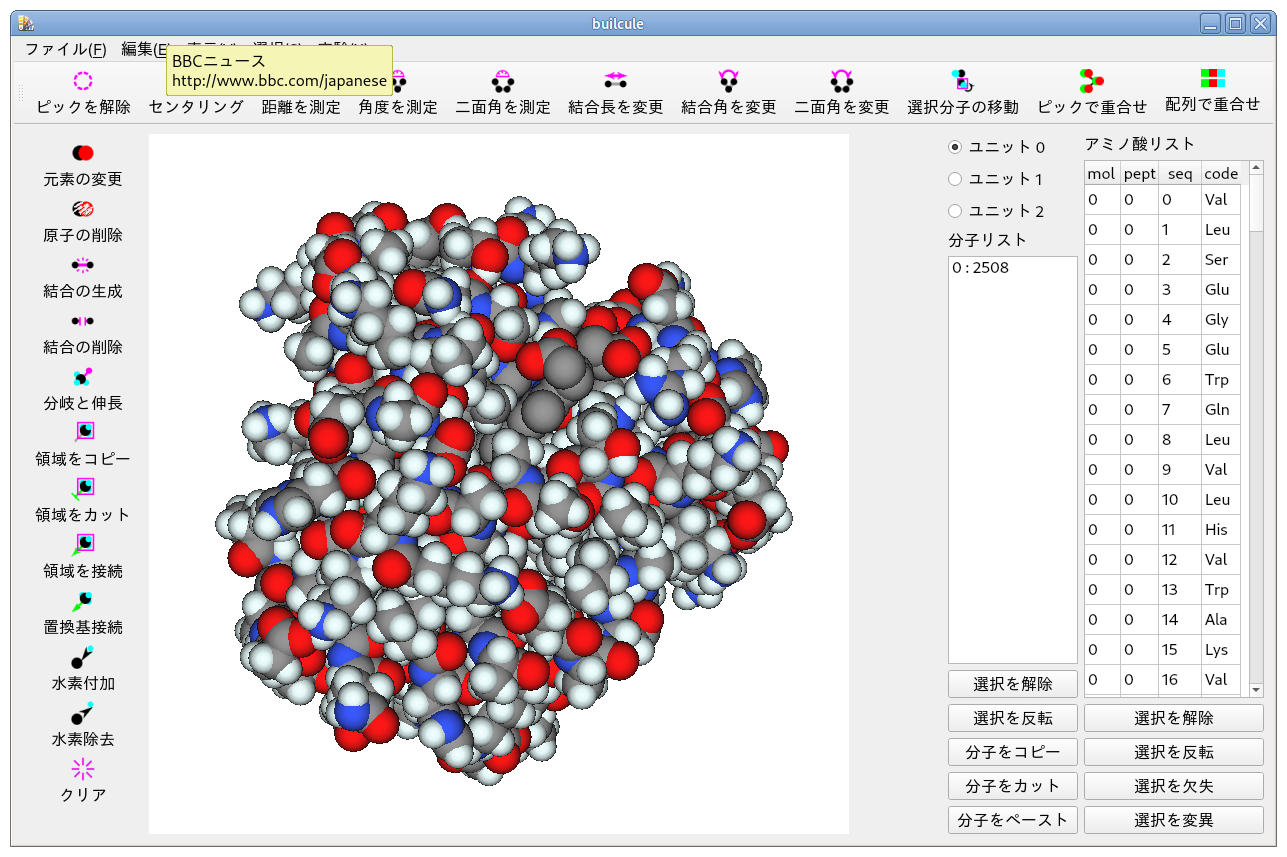
画像は上の状態に対し,[水素付加] を実行したところです.
薄い水色で示される水素が付加されています.
垂直ツールバーの![]() 水素除去ボタンをクリックすると上の状態になります.
水素除去ボタンをクリックすると上の状態になります.