タンパク質における共有結合の検知
原子の位置と元素からから共有結合を検知する基準を作成した.
まず.ファンデルワールス距離未満の炭素-炭素,炭素-窒素,炭素-酸素距離を測定してヒストグラムを作成して分析した.
次いで,共有結合距離と推定した数値のみでヒストグラムを作成して分析した.
これらの観察により,タンパク質での共有結合の性質を反映した結果が得られた.
この研究の目的は,PDB ファイルを開いたときに,タンパク質の共有結合を検知することである.
libbuilcule にはこの実験結果を取り入れ,「共有結合半径(文献値)の和 * 1.2 以下の距離であれば共有結合とみなす」としている.
インフォメーション
ソフトウェア
測定は Builcule を改変しておこなった.
目次(ページ内リンク)
共有結合半径よびファンデルワールス半径の文献値
方法
共有結合距離の測定
共有結合距離の分布
共有結合半径よびファンデルワールス半径の文献値
共有結合半径とファンデルワールス半径,二重結合距離については web 上で公開されていた数値を利用させていただいた.
これらの値を表にまとめた.共有合半径の和を「共有結合距離の期待値」とし,後で実測値と比較する.
| - | C | N | O |
|---|---|---|---|
| C | ファンデルワールス半径の和:3.40 共有合半径の和:1.54 二重結合:1.34 | ファンデルワールス半径の和:3.25 共有合半径の和:1.52 二重結合:1.27 | ファンデルワールス半径の和:3.22 共有合半径の和:1.50 二重結合:1.24 |
| N | - | ファンデルワールス半径の和:3.1 共有合半径の和:1.50 二重結合:1.20 | ファンデルワールス半径の和:3.07 共有合半径の和:1.48 二重結合:1.17 |
| O | - | - | ファンデルワールス半径の和:3.04 共有合半径の和:1.46 二重結合:1.14 |
試料と方法
試料は,手元にあったミオグロビン(1mbn)を利用した.
したがって,結果には,ヘムのデータが含まれる.
分岐限定法を使って,原子間距離がファンデルワールス半径の和以下となる組み合わせを抽出した.
そのなかの一部が共有結合している組み合わせである.
参考までに記しておく.
- タンパク質から,重複なしで原子を 2 個取り出した組み合わせを考える
- x 座標の差の絶対値を求め,それがファンデルワールス半径の和より小さければ以下の処理をおこなう(*)
- 原子間の距離を計算し,それがファンデルワールス半径の和より小さければ記録する
(*) ここが分岐限定法.x 座標の差がファンデルワールス半径の和より大きければ,それだけで共有結合ではありえない.全ての原子間距離を計算する手間が省略できる.
共有結合距離の測定
出力ファイル
出力ファイルの一部を示す.名前を 1mbn.csv とする
R の read.csv() 関数で開ける形式にしている.
C-S あるいは S-S の距離も測定できるようにしたが,ここでは使っていない.
CC,CN,CO,CS,SS 1.54272,1.40357,2.40624,2.71662,nan 1.55563,2.40624,1.22066,1.72916,nan 2.59615,2.41039,2.87924,1.71464,nan 2.61916,3.01496,3.10161,3.2619,nan
ファンデルワールス距離以下の原子間距離
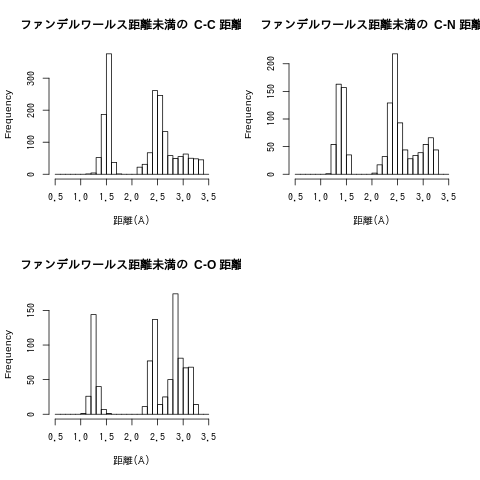
R を使い,出力されたデータでヒストグラムを描いた.
考察
原子間距離は,2.0Å 付近のデータを含まない領域を堺にして 2 つの領域に分布している.
ここでは 2.0Å 未満の距離を有するデータが共有結合距離として分析を進めた.
2.0Å 以上の距離を有するデータには,ファンデルワールス接触している原子と,1 原子挟んで隣接している原子(例えば,C-O-H といった構造の C と H)が含まれる.
ソースコード
covalent <- read.csv("1mbn.csv")
png("./covalent_0.png")
par(mfrow=c(2,2))
hist(covalent$CC, breaks=seq(0.5, 3.5, 0.1), xlab="距離(Å)", main="ファンデルワールス距離未満の C-C 距離")
hist(covalent$CN, breaks=seq(0.5, 3.5, 0.1), xlab="距離(Å)", main="ファンデルワールス距離未満の C-N 距離")
hist(covalent$CO, breaks=seq(0.5, 3.5, 0.1), xlab="距離(Å)", main="ファンデルワールス距離未満の C-O 距離")
dev.off()
共有結合距離の分布

2.0Å 未満のデータを抽出し,ヒストグラムを描いた(結合次数でピークが分裂するかどうかを調べるために,やや細かく範囲を区切っている).
考察
C-C 距離の分布で左側がなだらかなのは,二重結合も含まれるからであろう.
C-N 距離の分布で見て取れる大きな 2 個のピークは,ペプチド主鎖の C-N と N-CA 等の距離の違いに由来するのであろう.前者は二重結合性を有しているので距離が小さくなる.
C-O 距離の分布で右側に小さなピークが見られるが,Ser,Thr 側鎖の C-O 結合と推定する.
以上,タンパク質の構造を反映した結果が得られた.
ソースコード
covalent <- read.csv("1mbn.csv")
png("./covalent_1.png")
par(mfrow=c(2,2))
CC <- covalent$CC[covalent$CC < 2.0]
CN <- covalent$CN[covalent$CN < 2.0]
CO <- covalent$CO[covalent$CO < 2.0]
hist(CC, breaks=seq(1.0, 2.0, 0.01), xlab="距離(Å)", main="2.0Å未満の C-C 距離")
hist(CN, breaks=seq(1.0, 2.0, 0.01), xlab="距離(Å)", main="2.0Å未満の未満の C-N 距離")
hist(CO, breaks=seq(1.0, 2.0, 0.01), xlab="距離(Å)", main="2.0Å未満の未満の C-O 距離")
dev.off()
共有結合距離の上限値
共有結合距離を検知するためには,実測値の上限値を知っておく必要がある.
ここでは,共有結合距離の最大値 / 共有結合距離の期待値を計算した.
> max(CC)/1.54 [1] 1.135903 > max(CN, na.rm=TRUE)/1.52 [1] 1.031868 > max(CO, na.rm=TRUE)/1.50 [1] 1.00222
最初にまとめた共有結合距離の和よりやや大きな値となった.
この結果と Builcule によるモデリングの結果から,共有結合の検知範囲は共有結合距離の和 * 1.2 程度としておけばよさそうである.
必要に応じて係数を変更する必要がある.